Bayes_Poisson のバックアップ(No.6)
- バックアップ一覧
- 差分 を表示
- 現在との差分 を表示
- ソース を表示
- Bayes_Poisson へ行く。
授業で使います.
_ ポアソンの共役事前分布はガンマ分布とする
Gelman et al.: Bayesian Data Analysis, p.52, 2nd Edition.
ポアソン分布とガンマ分布の関係については繁桝算男『ベイズ統計入門』64ページを参照.
- ポアソン分布
#mimetex(p(y|\theta) = \frac{\theta^y \, e^{-\theta}}{y!})
- 尤度
#mimetex(\bf{y} = y_i, \dots , y_n)
#mimetex(p(\bf{y}|\theta) = l(\theta|\bf{y}) = \Pi \frac{1}{y_i!} \theta^{y_i} \, e^{-\theta})
#mimetex( \propto \theta^{t(y)} \, e^{-n \theta}) 指数属で表現すると
#mimetex( \propto e^{-n\theta}\, e^{t(y) log \theta} ) 文系学生相手にするので,指数法則を指摘する.
#mimetex(\theta^y = e^{log(\theta^y)} = e^{y log \theta}) 指数属とは関数が次の形で表されること(Lee: Bayesian Statistics, pp.60).
#mimetex(p(\bf{x}| \theta) = g(\bf{x}) \, h(\theta) \, e^{t(\bf{x}) \, \phi(\theta)}) 上の式では&mimetex(\frac{1}{y!});が消えているので注意
- 自然共役事前分布
#mimetex(p(\theta) \propto (e^{-\theta})^n e^{v log \theta} )
- 事後分布
#mimetex(\theta|\bf{y} \sim Gamma(\alpha + n \bar{y}, \beta + n))
- 医学関係ではポアソン分布を次のように書いている.
#mimetex(y_i \sim Poisson(x_i \theta)) &mimetex(y_i); と &mimetex(x_i); が観測数で,&mimetex(\theta); が未知のパラメータだが,&mimetex(x); を exposure,&mimetex(\theta); をrate と表現しており,分野違いの人間はいつまでたっても馴染みにくい.
_ R での実行例
- Albert Bayesian: Computation with R より
- 米国のある病院での心臓手術が失敗するケース数&mimetex(y); を考える.ここで失敗とは手術後30以内に死亡することである.これはポアソン分布に従うとする.手術(exposure)の総数を&mimetex(e);,exposureあたりの死亡率を&mimetex(\lambda); とすると,
- 米国のある病院での心臓手術が失敗するケース数&mimetex(y); を考える.ここで失敗とは手術後30以内に死亡することである.これはポアソン分布に従うとする.手術(exposure)の総数を&mimetex(e);,exposureあたりの死亡率を&mimetex(\lambda); とすると,
#mimetex(y \sim Poisson(e \lambda))
- &mimetex(\lambda);の最尤推定量は &mimetex( \bar{\lambda} = y/e);である.
- 上で述べたように事前共役分布はガンマ分布だが,二つのパラメータを設定する必要がある.ここでは,いま対象としている病院と同じ手術レベルの他の病院のデータを参考にして,&mimetex(\alpha = 16); と&mimetex(\beta = 15174); とする.すなわち事前分布は
#mimetex(\theta^{16-1} \, e^{-15174\, \theta} = Gamma(16,15174 ))
- すると事後分布は &mimetex(16 + y, 15174 + e); のガンマ分布となる.
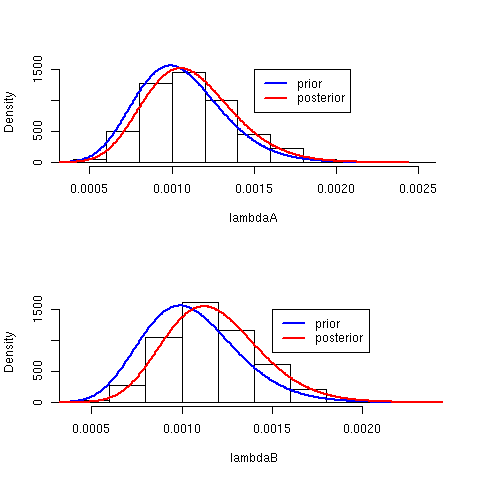
- さて,ここで新たに手術例が66件で,うち失敗が1件の病院と,手術例が1767件で,うち失敗が4件の病院があるとする.
my.alpha <- 16 my.beta <- 15174 lam <- my.alpha/my.beta lambdaA <- rgamma(1000, shape = my.alpha + 1, rate = my.beta + 66) lambdaB <- rgamma(1000, shape = my.alpha + 1767, rate = my.beta + 4) lambda <- seq(0, max(c(lambdaA, lambdaB)), length = 500)
par(mfrow = c(2,1))# , mar = rep(1, 4))
hist(lambdaA, freq = FALSE, main = "", ylim =c(0, 1600))
lines(lambda, dgamma(lambda, shape = my.alpha, my.beta),
col = "blue", lwd = 3)
lines(lambda, dgamma(lambda, shape = my.alpha+ 1,
my.beta + 66), col = "red", lwd = 3)
legend(0.0015, 1500, legend= c("prior", "posterior"),
col = c("blue","red"), lwd = 3)
hist(lambdaB, freq = FALSE, main = "", ylim =c(0, 1600))
lines(lambda, dgamma(lambda, shape = my.alpha, my.beta),
col = "blue", lwd = 3)
lines(lambda, dgamma(lambda, shape = my.alpha+ 4,
my.beta + 1767), col = "red", lwd = 3)
legend(0.0015, 1500, legend= c("prior", "posterior"),
col = c("blue","red"), lwd = 3)
_ 予測分布と負の二項分布
- (事前)予測密度関数&mimetex(f(x)); は,&mimetex(f(y|\lambda)); をサンプルのポアソン分布,&mimetex(g(\lambda)); を事前分布,&mimetex(g(y|\lambda)); を事後分布とすると,
#mimetex(f(y) = \frac{f(y|\lambda) \, g(\lambda)}{g(y|\lambda)})
#mimetex(p(y) = \frac{Poisson(y|\theta) Gamma(\theta|\alpha, \beta)}{Gamma(\theta|\alpha+y, 1 + \beta)})
#mimetex(=\frac{\Gamma (\alpha + y) \beta^\alpha}{\Gamma(\alpha)y! \, (1+\beta)^{\alpha+y}})
#mimetex(y \sim Neg-bin(\alpha, \beta)) これはポアソン分布の&mimetex(\theta); がガンマ分布に従う混合分布である.
_ ポアソン回帰
豊田秀樹『マルコフ連鎖モンテカルロ法』は良い本なんだけど,掲載サンプルではBRugsパッケージの利用を前提としている.ところがBRugsはCRANのパッケージから外されている.ポアソン回帰ならば,MCMCpackでも間に合う.
以下,『マルコフ連鎖モンテカルロ法』p.107掲載データ(生化学専攻大学院生の論文執筆数を性別,既婚・独身,子供の数で説明する)を glm と MCMCpoisson で実行する.
データ![]() MCMCpoisson.Rを読み込んで
MCMCpoisson.Rを読み込んで
library(MCMCpack)
source("MCMCpoisson.R")
posterior <- MCMCpoisson(y ~ x1 + x2 + x3)
plot(posterior)
summary(posterior)
Iterations = 1001:11000
Thinning interval = 1
Number of chains = 1
Sample size per chain = 10000
1. Empirical mean and standard deviation for each variable,
plus standard error of the mean:
Mean SD Naive SE Time-series SE
(Intercept) 0.6389 0.05432 0.0005432 0.002321
x1 -0.2852 0.05355 0.0005355 0.002204
x2 0.1300 0.06095 0.0006095 0.002382
x3 -0.1623 0.03891 0.0003891 0.001549
2. Quantiles for each variable:
2.5% 25% 50% 75% 97.5%
(Intercept) 0.5323 0.60324 0.6397 0.6746 0.74692
x1 -0.3908 -0.32266 -0.2837 -0.2498 -0.18105
x2 0.0082 0.09005 0.1311 0.1703 0.25447
x3 -0.2396 -0.18926 -0.1622 -0.1360 -0.08486
一般線形化モデル
pois.glm <- glm(y ~ x1 + x2 + x3,family=poisson())
summary(pois.glm)
Call:
glm(formula = y ~ x1 + x2 + x3, family = poisson())
Deviance Residuals:
Min 1Q Median 3Q Max
-2.0776 -1.6856 -0.3729 0.4936 6.9981
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) 0.63656 0.05457 11.665 < 2e-16 ***
x1 -0.28549 0.05433 -5.255 1.48e-07 ***
x2 0.13271 0.06092 2.179 0.0294 *
x3 -0.16118 0.03934 -4.097 4.19e-05 ***
(Dispersion parameter for poisson family taken to be 1)
Null deviance: 1817.4 on 914 degrees of freedom
Residual deviance: 1776.7 on 911 degrees of freedom
AIC: 3452.5
Number of Fisher Scoring iterations: 5
_ 参考文献
Gelman et al.: Bayesian Data Analysis, 2nd Edition,Chapman
Lee: Bayesian Statistics, Hodder Arnold
Albert Bayesian: Computation with R, Springer
繁桝算男『ベイズ統計入門』東京大学
豊田秀樹『マルコフ連鎖モンテカルロ法』朝倉書店
こういうページも参考に.